Un objectif fondamental de la biologie du développement est de comprendre les relations de lignée des cellules et des types de cellules les unes avec les autres, ainsi que les programmes moléculaires qui sous-tendent l'émergence de chaque type de cellule. In principle, developmental programs can be comprehensively described, as in Sulston and colleagues’ heroic reconstruction of the complete embryonic lineage of the roundworm Caenorhabditis elegans1.Cependant, C.elegans - instable, translucide et invariant du développement - est le seul organisme modèle pour lequel une telle description complète a été réalisée.
Since 2016, we and others have developed and applied new technologies for single-cell molecular profiling at the ‘whole-animal’ scale, including worm, fly, zebrafish, frog and mouse2,3,4,5,6,7.De telles études jettent les fondements des vues mondiales du développement animal, comme en remplissant la lignée de sulston de C. elegans with the gene expression programs of each cell type7,8.
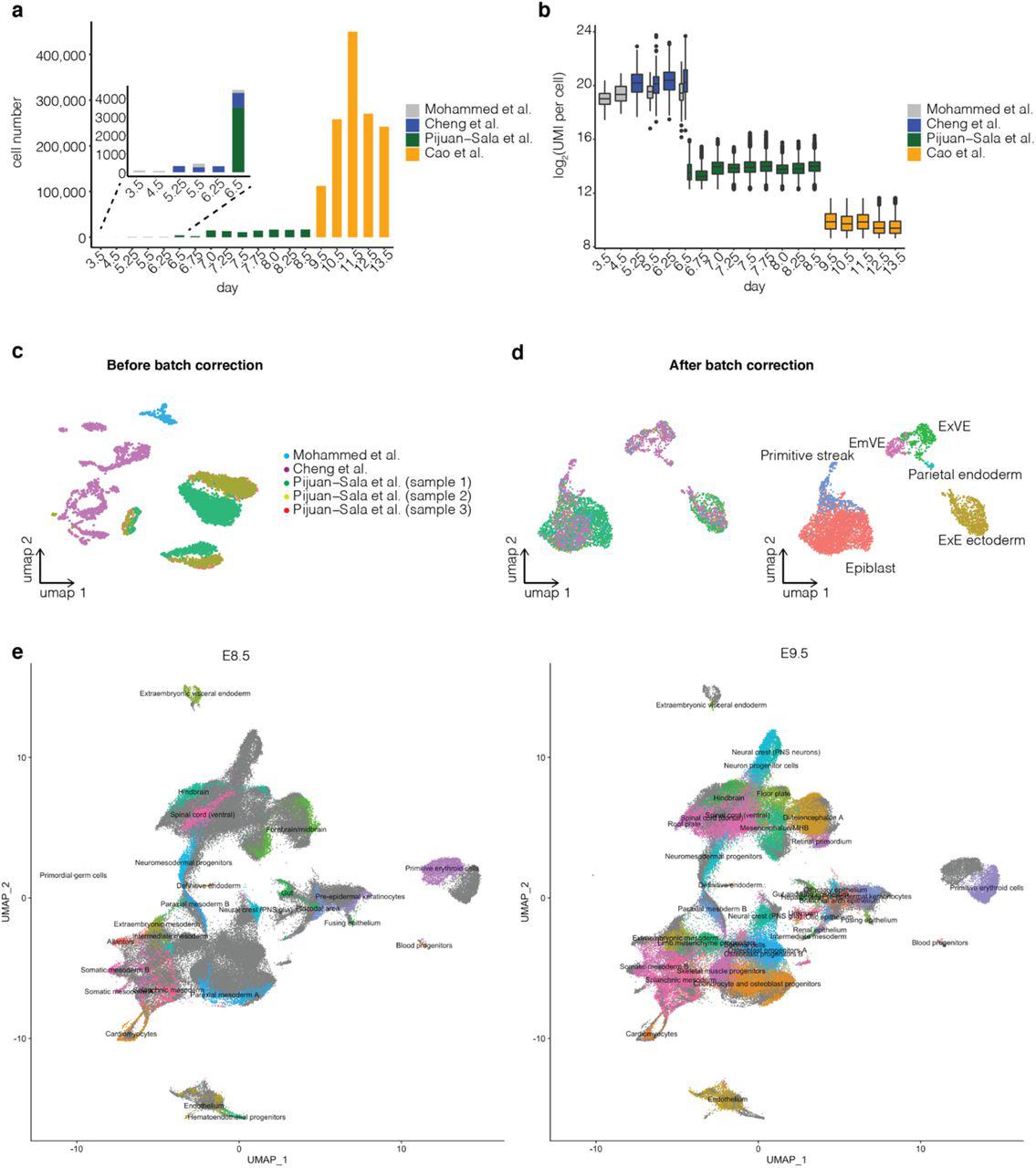
For mouse, the whole embryo has been profiled by scRNA-seq during implantation9,10, gastrulation2 and organogenesis4.Collectivement, ces études couvrent le développement à partir de dizaines de cellules de quelques types (E3.5) à des millions de cellules de centaines de types (E13.5).Cependant, les données associées n'ont pas encore été systématiquement intégrées d'une manière qui permet leur exploration robuste.Une telle intégration est difficile, à la fois pour des raisons techniques (e.g., différentes technologies et effets de lots) et en raison de la complexité du développement de la souris.
Ici, nous avons décidé de reconstruire systématiquement les principales trajectoires cellulaires de l'embryogenèse des mammifères de E3.5 à E13.5. Our primary strategy, inspired by Briggs and colleagues5, makes several assumptions: (1) although mouse development is variable, key patterns will be consistent across animals; (2) omnis cellula e cellula also applies to cell types (i.e., les types de cellules observés à un moment donné doivent provenir de types de cellules présents au moment précédent);(3) Nous échantillonnons fréquemment et suffisamment profondément pour que les types de cellules nouvellement détectés ne proviennent pas de types de cellules antécédents non détectés au moment précédent;et (4) en supposant que les points de temps d'échantillonnage sont étroitement espacés, la similitude transcriptionnelle est un moyen efficace de lier les types de cellules connexes à travers le temps.
Une prudence est que contrairement au sulston et al.La carte séminale de C de C. elegans, we focus here on reconstructing trajectories11, a concept related, but by no means equivalent, to lineage.Bien que ce soit une attente raisonnable que les cellules étroitement liées (e.g., siblings) will be transcriptionally similar8, the converse is not necessarily true. For example, lineally distant cells might be insufficiently divergent, or even convergent, obscuring lineage relationships12. Furthermore, even the expectation that closely related cells will be transcriptionally similar is not always met, as rapid changes can lead to ‘gaps’ in trajectories8.En somme, notre objectif ici est une feuille de route continue et navigable des états transcriptionnels des types de cellules pendant le développement de la souris.Une telle feuille de route peut contraindre les relations potentielles de lignée entre les types de cellules constituantes, mais il ne les spécifie pas explicitement.









